1/4
I. Introducción
En este artículo presentamos varios sistemas, basados en métodos de aprendizaje computacional, con métodos estadísticos, para el análisis de imágenes de ultrasonido (US) médico en 2 y 3 dimensiones, con aplicaciones en: ecocardiografía, salud materno-fetal y oncología. Todos los sistemas son resultados de la colaboración de dos instituciones de educación superior (Universidad Nacional Autónoma de México y Universidad Autónoma Metropolitana) y una institución de salud pública (Instituto Nacional de Perinatología), en México.
El ultrasonido médico ofrece varias ventajas con respecto a otras modalidades de imágenes médicas (por ejemplo, rayos X, tomografía computarizada y resonancia magnética). Los equipos tienen dimensiones reducidas y la adquisición de imágenes es muy versátil. Un experto puede explorar diferentes regiones anatómicas con facilidad. No se utiliza radiación ionizante para formar la imagen –como es el caso con rayos X-, por lo que es posible adquirir imágenes de ultrasonido de cualquier paciente con toda seguridad. Los costos de los equipos son los más bajos de todas las modalidades de imágenes médicas disponibles. Las principales desventajas de las imágenes médicas de ultrasonido, son el alto contenido de ruido de moteado (speckle) [1] y la necesidad de que un experto médico realice simultáneamente la adquisición de imágenes y la interpretación en el paciente (es decir, es una modalidad de imágenes médicas que depende del usuario).
La penetración del ultrasonido en el cuerpo humano, para la formación de imágenes, está limitada a unos 15 cm, que permiten todavía visualizar una buena parte de la anatomía humana. Los avances continuos en la adquisición, el procesamiento de señales y la formación de imágenes, han aumentado constantemente la calidad, reduciendo los niveles de ruido speckle. [2] Actualmente, el ultrasonido es la modalidad más utilizada en medicina, [3] y nos permite, en algunos casos, visualizar estructuras de tejido blando que no se pueden ver con rayos X. Por ejemplo, los tumores de mama en mujeres jóvenes pueden muchas veces detectarse mejor con ultrasonido que con rayos X (mastografía). Los equipos modernos permiten visualizar imágenes (2D) o volúmenes (3D), estáticos o como secuencias de video, de la anatomía del paciente.
Por sus altos niveles de seguridad y accesibilidad, el ultrasonido se ha utilizado activamente en el monitoreo de la salud materno fetal durante la gestación. También se utiliza mucho en ecocardiografía (fetal y posnatal en todas las edades), hígado, próstata, músculos, vascularidades y recientemente para detectar la superficie de algún hueso de interés durante cirugías ortopédicas. En este artículo presentamos varios sistemas de análisis de imágenes de ultrasonido médico, desarrollados en colaboraciones entre la Facultad de Ingeniería y el IIMAS de la UNAM, la UAM Unidad Iztapalapa y el Instituto Nacional de Perinatología (INPER) en la Ciudad de México. Los sistemas se basan en técnicas de aprendizaje computacional (machine learning), para realizar automáticamente alguna tarea de apoyo al diagnóstico médico en cardiología, perinatología (salud materno-fetal) y oncología de mama, como se describe a continuación.
2/5
II. Anotación automática del ventrículo izquierdo
Uno de los índices más importantes del buen funcionamiento del corazón es la fracción de eyección, calculada como el volumen de sangre que expulsa el ventrículo en cada contracción. La fracción de eyección se mide utilizando imágenes de ecocardiografía (ultrasonido) del ventrículo izquierdo (figura 1). Para una medición exacta, es necesario anotar el contorno del ventrículo cuando tiene el máximo volumen de sangre (diástole) y cuando tiene el volumen más pequeño (sístole).
En un trabajo de colaboración entre el Laboratorio Avanzado de Procesamiento de Imágenes de la Facultad de Ingeniería y la Unidad Yucatán del IIMAS de la UNAM, desarrollamos un sistema automático para la anotación del ventrículo izquierdo en imágenes de ultrasonido, que nos permitirá el cálculo automatizado de la fracción de eyección. A continuación, describimos el método de modelado estadístico, de las formas que puede tomar un órgano, que utilizamos para la anotación automática del ventrículo izquierdo.
II.1. Modelo de distribución de puntos y modelo de formas activas (active shape model) del ventrículo izquierdo
Un modelo de distribución de puntos (MDP) [4] es una manera efectiva de modelar matemáticamente la variedad de formas que puede tomar el contorno de uno o varios órganos en 2D (imágenes) y en 3D (volúmenes). El modelo se construye a partir de un conjunto de ejemplos de las formas de interés. Sobre el contorno o la superficie de cada ejemplo, se muestrea un número (n) de puntos de referencia (landmarks), como se ilustra en la figura 2, con ejemplos de formas del ventrículo izquierdo.
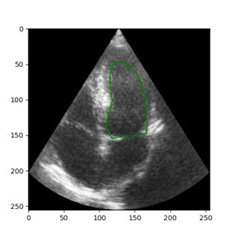
Figura 1. Ultrasonido del ventrículo izquierdo con la anotación del experto
Las coordenadas (x,y) de todos los puntos de referencia de cada ejemplo de entrenamiento, se almacenan en un vector. Todos los ejemplos se alinean dentro del mismo sistema de coordenadas de referencia, como se describe en la referencia 4. Con todos los vectores alineados del conjunto de entrenamiento, podemos fácilmente calcular la forma promedio como el vector promedio, como se ilustra en la figura 2.
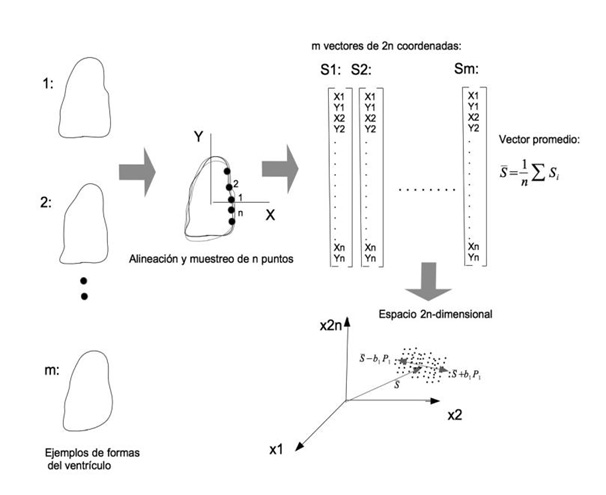
Figura 2. Construcción de un modelo de distribución de puntos (MDP) del ventrículo izquierdo
Así como la representación gráfica de un vector de tres coordenadas (x,y,z) corresponde a un punto en un sistema de 3 dimensiones (x,y,z), la misma interpretación se puede aplicar a vectores con cualquier número de coordenadas: un vector de n coordenadas corresponde a un punto en un espacio de n dimensiones. Nuestra forma promedio calculada anteriormente, corresponde a un punto en el espacio de 2n-dimensiones (n puntos con dos coordenadas x,y), como se ilustra en la figura 2. Como todos los vectores de nuestros ejemplos del ventrículo son similares, esperamos que los puntos correspondientes ocupen una región compacta en el espacio de 2n-dimensiones. Podemos entonces modelar la forma aproximada y el tamaño de esta región, con un análisis de componentes principales (ACP) de todos los vectores, como se describe en la referencia 4, y podemos generar nuevas formas del ventrículo como la suma del vector (ventrículo) promedio ŝ más un incremento b alrededor del promedio, dentro de la región de “ventrículos” que determinamos con el ACP, como se ilustra en la figura 2 para un incremento b1P1, donde b1 es un escalar y P1 es un vector (abajo a la derecha en la figura 2). Las formas correspondientes se muestran junto con la forma promedio del ventrículo en la figura 3.
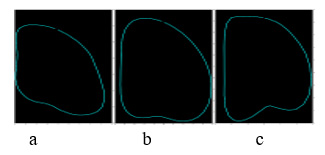
Figura 3. Formas del MDP del ventrículo izquierdo para: a) s=ŝ - b1P1; b)s=ŝ; c)s=ŝ + b1P1
Cootes y Taylor [4] desarrollaron también un método de ajuste automático del MDP a una nueva imagen, que bautizaron como modelo de formas activas (active shape model). El método de ajuste se basa en la búsqueda de bordes cercanos a la forma inicial del MDP. Con la detección de bordes se ajusta la forma del MDP y se repite la búsqueda y el ajuste de un número fijo de iteraciones. Usualmente, basta con 50 iteraciones para lograr un buen ajuste. En la figura 4 mostramos 3 ejemplos de resultados del ajuste automático del MDP del ventrículo izquierdo, entrenado con 50 ejemplos.
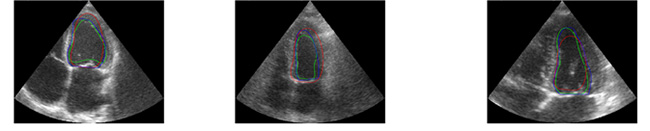
Figura 4. Resultados de la anotación automática del ventrículo izquierdo, en tres imágenes de validación: rojo, inicialización de la forma promedio; azul, la anotación del experto, y verde, la segmentación automática final.
III. Ultrasonido del Cerebro Fetal
Como hemos mencionado, el ultrasonido es una técnica no invasiva, que trabaja en tiempo real, no requiere el uso de radiación ionizante, el equipo es compacto y portátil, el transductor es pequeño y fácilmente manipulable. Estas cualidades hacen que sea la modalidad más utilizada en obstetricia, para la valoración de la salud materno-fetal. En particular, el estudio del cerebro fetal mediante imágenes de ultrasonido (US), puede ayudar a los expertos obstetras a dar un seguimiento del neurodesarrollo del feto. Sin embargo, la ecografía presenta algunas limitaciones que pueden restringir la adecuada medición de diversos índices y, como consecuencia, dificultar un buen diagnóstico y valoración de la salud fetal. El ultrasonido presenta variaciones entre pacientes y entre equipos; las imágenes no tienen contornos bien definidos y muestran ruido multiplicativo (speckle) con variaciones en intensidad, y puede presentar diversos artefactos y sombras acústicas. [5] A continuación, presentamos tres sistemas de análisis de imágenes de US del cerebro fetal –basados en métodos de aprendizaje computacional-, que contribuyen al diagnóstico clínico exacto y eficiente.
III.1. Medición automática del pliegue nucal
Durante la gestación, el pliegue nucal se forma por la acumulación normal de líquido en la base del cráneo del feto, sin embargo, un grosor fuera de rangos normales del pliegue nucal es el principal indicador para la detección de síndrome de Down, durante el segundo trimestre de la gestación. En esta sección presentamos la construcción de un modelo de formas activas (active shape model), para la medición automática del grosor del pliegue nucal, en imágenes de ultrasonido del cerebro fetal.
III.1.1. Modelo de distribución de puntos y modelo de formas activas (active shape model) del pliegue nucal
Un modelo de distribución de puntos puede también representar las formas de varios objetos. En la figura 5 mostramos los ejemplos de entrenamiento y la forma promedio de todas las estructuras anatómicas de interés durante la medición del pliegue nucal –línea media, cerebelo, placa occipital y borde de la piel–, con los puntos muestreados correspondientes, que permitirán la construcción del MDP del pliegue nucal. Los vectores de entrenamiento se formaron con las coordenadas (x,y) de todos los puntos de las 4 estructuras anatómicas que se ilustran en la figura 5.
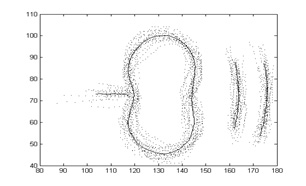
Figura 5. Forma promedio de las estructuras del pliegue nucal (línea continua) y los puntos muestreados de 20 ejemplos de entrenamiento.
En la figura 6 mostramos cómo se deforma el MDP de la “plantilla del pliegue nucal”.
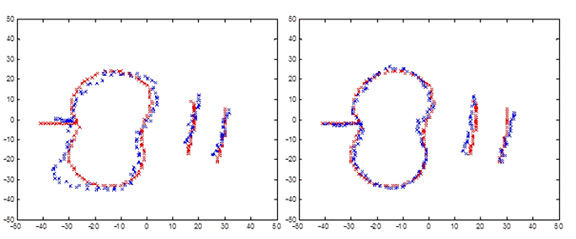
Figura 6. Formas del MDP del pliegue nucal para: a) s=ŝ - b1P1; b) s=ŝ + b1P1; se muestra en rojo la forma promedio y en azul la nueva forma
En la figura 7 mostramos dos ejemplos del resultado del ajuste automático del MDP del pliegue nucal, a partir de una forma inicial.
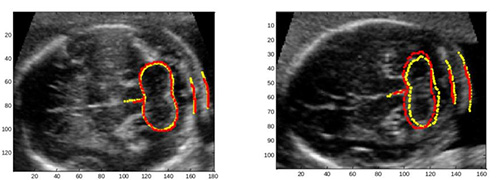
Figura 7. Resultados del ajuste automático del ASM del pliegue nucal: rojo, anotación experta; amarillo, ASM.
III. 2. Anotación automática del cerebelo en 3 dimensiones (3D)
2. Anotación automática del cerebelo en 3 dimensiones (3D) El cerebelo es una estructura cerebral importante, por su participación en las funciones cognitivas, como la atención y el lenguaje, así como la coordinación de movimientos, precisión y sincronización precisa. [6] También se puede utilizar para evaluar la edad gestacional o afecciones prenatales, como las anomalías en la fosa posterior craneal y defectos del tubo neural. [6,7] La segmentación automática de estructuras cerebrales en la modalidad de ultrasonido, es una tarea desafiante debido a los artefactos, como el “speckle” y la poca homogeneidad de intensidad, debido a los movimientos fetales durante los procesos de adquisición de imágenes.
Para la segmentación automática del cerebelo en 3D, una experta en perinatología realizó la anotación manual de los 15 principales cortes que conforman el cerebelo (planos axiales), en 20 volúmenes de ultrasonido, con el fin de generar posteriormente un modelo de distribución de puntos de la forma del cerebelo en tres dimensiones (3D) y sus modos de variación, que nos permiten un buen ajuste en un nuevo volumen. [8] Un MDP de formas en tres dimensiones se construye de manera análoga a un modelo de formas en 2 dimensiones, como lo presentamos en la figura 1. Sin embargo, ahora se muestrean puntos sobre la superficie de cada ejemplo de forma 3D y se almacenan las coordenadas (x,y,z) de todos los puntos en un vector. La forma promedio y los modos de variación de forma, se calculan con el mismo análisis de componentes principales, descrito anteriormente para el MDP del ventrículo izquierdo (figura 8).
El ajuste del MDP 3D se basó en una función objetivo, que depende de los parámetros de forma y postura (ángulo, escala y traslaciones) del MDP, para la detección de bordes que corresponden al cerebelo, dentro de un volumen de ultrasonido del cerebro fetal. En nuestros resultados observamos que la anotación automática del cerebelo -utilizando el MDP 3D- produce en promedio un 90% de correspondencia con los volúmenes anotados por una experta, como se ilustra en la figura 9, donde se muestra en amarillo la anotación tridimensional de la experta y en azul la anotación automática.
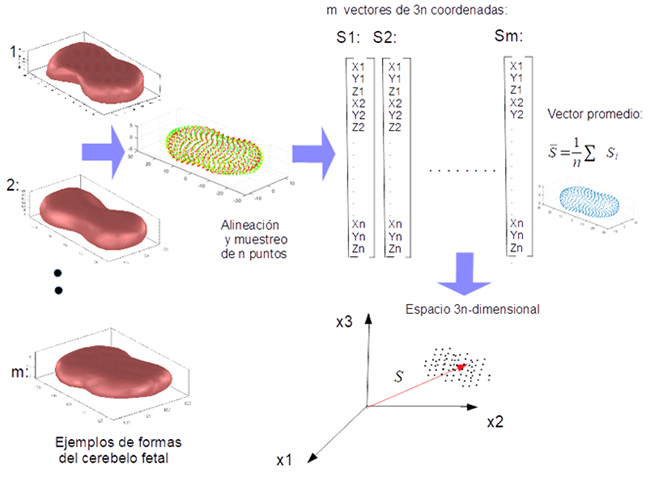
Figura 8. Construcción del MDP tridimensional del cerebelo fetal
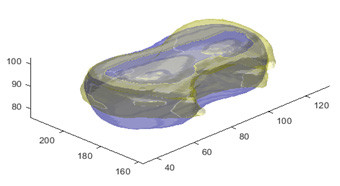
Figura 9. Comparativa de la segmentación de la experta (amarillo) contra la automática (azul)
III.3. Composición espacial de imágenes cerebrales fetales de US 3D
En el caso particular de la neuroecografía fetal, existe un problema de oclusión causada por la calcificación y el endurecimiento craneal durante el segundo y el tercer trimestre gestacional, lo que impide el paso del sonido al interior del cráneo. Esto repercute en la aparición de sombras en las regiones correspondientes al cerebro fetal. [9,10] Este fenómeno impide la correcta valoración del cerebro, así como la medición de diversas estructuras internas. En la figura 10 se muestran dos ejemplos de estudios cerebrales fetales con sombras acústicas. Se puede notar claramente la ausencia de información correspondiente al tejido cerebral.Para lidiar con el problema de las oclusiones acústicas cerebrales, se ha propuesto una estrategia basada en algoritmos de procesamiento digital de imágenes y aprendizaje computacional. En la figura 11 se muestra la metodología propuesta, con el objetivo de mejorar la calidad de las imágenes cerebrales fetales de US en 3D, disminuyendo la cantidad de sombras acústicas.
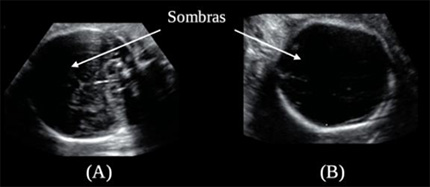
Figura 10. Ejemplos de imágenes de US fetal cerebral con artefactos de oclusión acústica
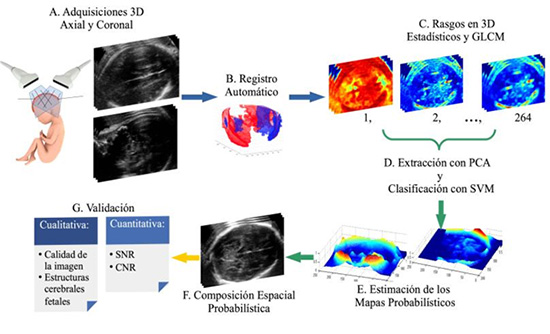
Figura 11. Diagrama general de la metodología propuesta para la composición de estudios cerebrales fetales de US 3D [7]
La metodología comienza con la adquisición de dos o más estudios de US 3D de la cabeza fetal. El propósito consiste en hacer la composición o fusión de varios estudios de US, para generar uno nuevo de mejor calidad y menor cantidad de sombras acústicas. De acuerdo con lo reportado por Timor-Trisch et al., [9] diferentes ángulos de adquisición pueden brindar información complementaria del tejido cerebral. Por lo tanto, la metodología comienza con la adquisición de estudios cerebrales desde dos ángulos: axial y coronal, como se puede observar en la figura 11A. El siguiente paso es alinear ambos volúmenes en un mismo plano de referencia, para lo que se propuso un algoritmo de alineación o registro automático basado en bosques aleatorios (figura 11.B). [11] Para hacer una adecuada composición o fusión de los estudios cerebrales, se propuso estimar un factor de ponderación. Este factor tiene como objetivo asignar valores cercanos a “1” a las regiones de la imagen que contengan información del tejido cerebral y valores cercanos a “0” a las regiones que contengan sombras acústicas. Para lograr esto, se propone estimar mapas probabilísticos para cada volumen de US a fusionar. Estos mapas probabilísticos fueron obtenidos mediante una máquina de soporte vectorial (MSV), alimentada con un conjunto de 264 mapas de textura llamados matrices de coocurrencia de escala de grises (MCEG), los cuales fueron seleccionados con un análisis de componentes principales (ACP) (figuras 11C y 11D). La MSV es un algoritmo de aprendizaje computacional, el cual fue entrenado para clasificar y ponderar regiones con tejido cerebral y zonas con oclusiones acústicas, generando así los mapas de ponderación (figura 11.E). Para hacer la fusión de los diversos volúmenes, se utilizó la norma euclidiana ponderada con los mapas previamente obtenidos. Como resultado, se obtiene un nuevo estudio cerebral fetal de US 3D, que contiene la información de los estudios fusionados y ponderados de forma probabilística (figura 11.F).
Finalmente, para evaluar el desempeño, medimos la relación señal a ruido (SNR, signal to noise ratio) y la relación contraste a ruido (CNR, contrast to noise ratio), las cuales mejoraron de 7.8+/-2.8 a 10.5+/-2.7 para SNR y de 5.1+/-1.9 a 12.1+/-2.3 para CNR. Esto refleja cuantitativamente la mejoría en la calidad del nuevo estudio generado. Además, expertos en obstetricia efectuaron una evaluación cualitativa, quienes mencionan que estructuras como la línea media (LM), cerebelo (CER) o circunferencia craneal (CC), se pueden apreciar con mejor calidad en el estudio de US, resultado de la composición. En la figura 12 se muestra la comparación entre un estudio tradicional y el resultado del método propuesto. En la figura 12.A se observan algunas sombras acústicas. En contraste, en la figura 12.B se puede observar una mayor cantidad de tejido cerebral, así como una mejor calidad en diversas estructuras.
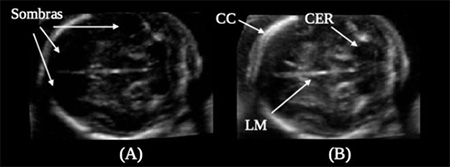
Figura 12. Ejemplo de una imagen de US fetal cerebral sin composición (A) y con el algoritmo de composición propuesto (B). CC: circunferencia craneal, CER: cerebelo y LM: línea media
Se considera que el algoritmo propuesto puede ayudar a generar un nuevo estudio cerebral fetal de US 3D de mejor calidad, que permita a los expertos en obstetricia efectuar una mejor valoración del cerebro, así como facilitar las mediciones de diversas estructuras, como el cerebelo o la circunferencia craneal. Como trabajo a futuro, se pretende seguir explorando nuevos algoritmos basados en redes neuronales artificiales, de los cuales ya se tienen resultados preliminares. [12]
3/5
IV. Anotación automática de tumores de mama en ultrasonido 2D
El cáncer de mama se ha convertido en la causa número uno de muertes en mujeres alrededor del mundo. La detección temprana y la estimación de riesgo son críticas para mejorar el pronóstico del paciente. Aunque la resonancia magnética es la herramienta recomendada por la OMS para la visualización del cáncer de mama, el ultrasonido se ha convertido en una herramienta muy útil para asistir y complementar este procedimiento, principalmente en mujeres jóvenes, en las cuales los tumores tienden a ser más grandes y malignos, ya que es capaz de detectar las lesiones en tejido mamario denso, usualmente presente en mujeres menores a 30 años, con mayor sensibilidad que la mamografía. Otra ventaja es que es la mejor herramienta para distinguir entre masas sólidas, como fibroadenomas benignos y cáncer, y quistes benignos llenos de líquido, dando indicios de la malignidad de la masa. El ultrasonido puede ser utilizado también para guiar procedimientos quirúrgicos, como las biopsias de tumores de mama, procedimiento que es más exacto y confiable para el diagnóstico de cáncer. [13]
Se han realizado diversos esfuerzos para efectuar la segmentación automática de lesiones en imágenes de ultrasonido, con la finalidad de localizar y delinear los contornos, y así ayudar a los médicos en la visualización, la detección y el diagnóstico de cáncer. Dentro de los métodos propuestos destacan los basados en el aprendizaje computacional. Estos métodos tienden a ser más robustos, ya que toman en cuenta más de una característica para la identificar lesiones. Métodos supervisados como redes Neuronales Artificiales, AdaBoost y Random Forest, son comúnmente utilizados para la clasificación de pixeles y así poder realizar la diferenciación del tejido lesionado y el tejido adyacente. [14, 15] Sin embargo, las características extraídas de la imagen y los parámetros de entrenamiento deben ser cuidadosamente elegidos y optimizados para obtener el mejor modelo de aprendizaje posible. El patrón interno de eco tiene mayor poder descriptivo que el nivel de intensidad de gris. Diferentes descriptores de textura de primer orden (momentos centrales del histograma) [16], segundo orden (descriptores basados en matrices de coocurrencia y run-length) [17] y otros (transformada de Fourier y Hermite), [18] han sido utilizados para caracterizar las texturas de las lesiones de mama en imágenes de ultrasonido. En la correcta selección de características y la reducción de dimensiones para la optimización de los resultados en la clasificación, se han propuesto diversos métodos como el Análisis de Componentes Principales (ACP) y la optimización genética. [19]
Aunque los métodos de aprendizaje computacional son robustos, usualmente no pueden producir segmentaciones exactas y el refinamiento de la segmentación, como la eliminación de regiones falso-positivas, es necesaria. [20] Imágenes de probabilidad, creadas a partir de los resultados en la estimación de probabilidad de clase, obtenida mediante los métodos de aprendizaje, pueden ser utilizadas para la eliminación de regiones falso-positivas y desconectar regiones débilmente conectadas. En la figura 13 se presenta una metodología completa para la segmentación de lesiones de mama en imágenes de ultrasonido, incluyendo la selección de características y parámetros óptimos, para entrenar modelos de aprendizaje computacional, optimizado con un algoritmo genético simple y el posprocesamiento necesario para la eliminación de regiones falso-positivas. Todas las etapas del método se entrenaron con la validación cruzada en 88 imágenes con k = 4 (25% de imágenes de validación).
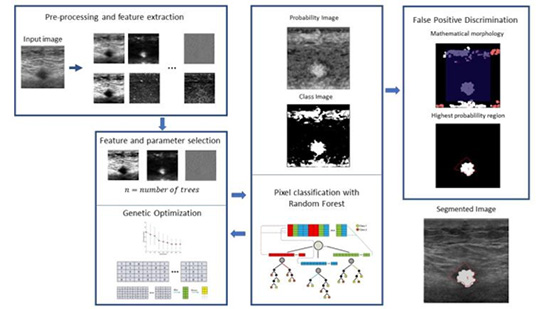
Figura 13. Metodología de segmentación de lesiones de mama en imágenes de ultrasonidos
Esta metodología incluye la extracción de características de textura de primer y segundo orden, así como la selección de características, la reducción de dimensión y la selección de parámetros óptimos. Posteriormente, se realiza la eliminación de regiones falso-positivas, mediante el uso de la imagen de probabilidad.
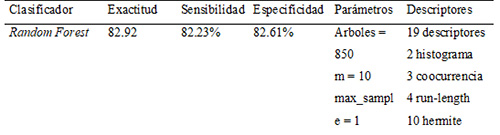
En la tabla 1 se presenta el modelo con los mejores resultados de exactitud, sensibilidad y especificidad, y los parámetros y descriptores de textura seleccionados por el algoritmo genético para la optimización de los resultados.
Tabla 1.Resultados de la segmentación mediante optimización genética
4/5
V. Comentarios finales
En este artículo presentamos 3 sistemas, basados en técnicas de aprendizaje computacional, para la anotación automática de estructuras anatómicas, como: el ventrículo izquierdo, el pliegue nucal y el cerebelo. Presentamos la aplicación de un método de modelado estadístico de formas: active shape model en 2D –ventrículo y pliegue nucal- y en 3D -cerebelo-. El método modela (o aprende) la variedad de formas que pueden tomar las estructuras anatómicas, a partir de un conjunto de ejemplos. Presentamos también un sistema de mejora de la calidad de las imágenes de ultrasonido del cerebro fetal, que fusiona varias vistas de la misma región con un método de aprendizaje probabilístico, logrando así una mejor visualización de las estructuras cerebrales sin las sombras que produce el cráneo del feto en desarrollo. Finalmente, presentamos también un sistema para la anotación automática de tumores de mama, basado en la extracción de características de textura de primer y segundo orden, así como la selección de características, la reducción de dimensión y la selección de parámetros óptimos. A través del cálculo de una imagen de probabilidad, se estima con buena exactitud la región que ocupa el tumor en la imagen de ultrasonido.
Todos los sistemas reportados en este artículo están concebidos para apoyar a los especialistas en el diagnóstico y la toma de decisiones clínicas. El tipo de métodos basados en modelos estadísticos de formas y texturas permite la predicción, con buena exactitud, a partir de algunas decenas de ejemplos de entrenamiento. Esto a su vez hace viable la mejora de la calidad de los diagnósticos médicos con ultrasonido, sin el requerimiento de grandes volúmenes de imágenes de entrenamiento de los métodos de análisis basados en redes neuronales profundas. La combinación de las dos aproximaciones tiene potencial para hacer más efectivo el diagnóstico clínico con imágenes de ultrasonido en 2D y 3D, fijas y en movimiento.
Agradecimientos
Agradecemos el apoyo de la UNAM a través de los proyectos PAPIIT TA101121 y PAPIIT IV100420. Así como el apoyo de la SECTEI de la Cd. de México a través del proyecto SECTEI/202/2019.
5/5
Bibliografía
[1] Y. Yu & S. T. Acton, “Speckle reducing anisotropic diffusion,” en IEEE Transactions on image processing, vol. 11, no. 11, pp.1260-1270, 2002.
[2] K. Y. Leung, “Applications of Advanced Ultrasound Technology in Obstetrics,” en Diagnostics, vol. 11, no. 7, p. 1217, 2021.
[3] A. L. Klibanov & J. A. Hossack, “Ultrasound in radiology: from anatomic, functional, molecular imaging to drug delivery and image-guided therapy,” en Investigative radiology, vol. 50, no. 9, p. 657, 2015.
[4] T. Cootes, C. Taylor, D. Cooper, et al., “Active shape models-Their training and application”, en Computer Vision and Image Understanding, vol. 61, no. 1, pp. 38-59, 1995.
[5] S. H. Contreras Ortiz, T. Chiu, and M. D. Fox, “Ultrasound image enhancement: A review,” en Biomedical Signal Processing and Control, vol. 7, pp. 419–428, Sep 2012.
[6] F. Liu, Z. Zhang, X. Lin, et al., “Development of the human fetal cerebellum in the second trimester: a postmortem magnetic resonance imaging evaluation,” en Journal of anatomy, vol. 219, no. 5, pp. 582-588, 2011.
[7] M. R. Hatab, S. W. Kamourieh, & D. M. Twickler, “MR volume of the fetal cerebellum in relation to growth,” en Journal of Magnetic Resonance Imaging: An Official Journal of the International Society for Magnetic Resonance in Medicine, vol. 27, no. 4, pp. 840-845, 2008.
[8] G. Velásquez-Rodríguez, F. Arámbula-Cosío, & Boris-Escalante, “Automatic segmentation of the fetal cerebellum using spherical harmonics and gray level profiles,” en 11th International Symposium on Medical Information Processing and Analysis, vol. 9681, International Society for Optics and Photonics, 2015.
[9] I. Timor-Trisch, A. Monteagudo, and H. Cohen, Neuroecografia Prenatal y Neonatal, vol. 1. MARBAN, 2004.
[10] G. Kossoff, K. Griffiths, and C. Dixon, “Is the quality of transvaginal images superior to transabdominal ones under matched conditions?,” Ultrasound Obstet Gynecol, vol. 1, pp. 29–35, Jan 1991.
[11] J. Perez-Gonzalez, F. Arámbula-Cosío, J.C. Huegel, et al., “Probabilistic Learning Coherent Point Drift for 3D Ultrasound Fetal Head Registration,” en Computational and Mathematical Methods in Medicine, vol. 2020, pp. 1-14. https://doi.org/10.1155/2020/4271519.
[12] J. Perez-Gonzalez, N. Hevia Montiel, V. Medina-Bañuelos, “Deep Learning Spatial Compounding from Multiple Fetal Head Ultrasound Acquisitions” en International Workshop on Preterm, Perinatal and Paediatric Image Analysis, Held in Conjunction with MICCAI 2020, Lima Perú. Lecture Notes in Computer Science, vol. 12437. Springer, Cham. https://doi.org/10.1007/978-3-030-60334-2_30.
[13] J. Olveres, et al., “What is new in computer vision and artificial intelligence in medical image analysis applications,” en Quantitative Imaging in Medicine and Surgery, vol. 11, no. 8, pp. 3830853–3833853, Aug. 2021, doi: 10.21037/QIMS-20-1151.
[14] P. Jiang, J. Peng, G. Zhang, et al., “Learning-based automatic breast tumor detection and segmentation in ultrasound images,” 2012 9th IEEE International Symposium on Biomedical Imaging (ISBI), May 2012, pp. 1587–1590, doi: 10.1109/ISBI.2012.6235878.
[15] F. Torres, B. Escalante-Ramirez, J. Olveres, et al., “Lesion Detection in Breast Ultrasound Images Using a Machine Learning Approach and Genetic Optimization,” In: Morales A., Fierrez J., Sánchez J., Ribeiro B. (eds) Pattern Recognition and Image Analysis. IbPRIA 2019. Lecture Notes in Computer Science, vol 11867. Springer, Cham. https://doi.org/10.1007/978-3-030-31332-6_26
[16] A. Madabhushi and D. N. Metaxas, “Combining low-, high-level and empirical domain knowledge for automated segmentation of ultrasonic breast lesions,” en IEEE Transactions on Medical Imaging, vol. 22, no. 2, pp. 155–169, 2003.
[17] B. Liu, H. D. Cheng, J. Huang, et al., “Fully automatic and segmentation-robust classification of breast tumors based on local texture analysis of ultrasound images,” en Pattern Recognition, vol. 43, no. 1, pp. 280–298, Jan. 2010, doi: 10.1016/J.PATCOG.2009.06.002.
[18] F. Torres, Z. Fanti, and F. Arámbula-Cosío, “3D freehand ultrasound for medical assistance in diagnosis and treatment of breast cancer: Preliminary results,” in Proceedings of SPIE - The International Society for Optical Engineering, 2013, vol. 8922, doi: 10.1117/12.2041806.
[19] C. Munoz-Meza and W. Gomez, “A feature selection methodology for breast ultrasound classification,” en 2013 10th International Conference on Electrical Engineering, Computing Science and Automatic Control, CCE 2013, pp. 245–249, 2013, doi: 10.1109/ICEEE.2013.6676056.
[20] J. Shan, H. D. Cheng, and Y. Wang, “A novel automatic seed point selection algorithm for breast ultrasound images,” in 2008 19th International Conference on Pattern Recognition, Dec. 2008, pp. 1–4, doi: 10.1109/ICPR.2008.4761336.












